博客
使用 cuCIM 和 NVIDIA GPU DIRECT STORAGE加速数字病理学工作流程
作者:Greg Lee、Rahul Choudhury 和 Gigon Bae
转自:英伟达开发者博客
全玻片成像(WSI)是使用全玻片扫描仪对载玻片上的组织进行数字化,在医疗保健领域越来越受欢迎。WSI 使组织病理学、免疫组织化学和细胞学的临床医生能够:
|
这篇文章解释了 GPU 加速工具包如何提高输入/输出 (I/O) 性能和图像处理任务。更具体地说,它详细介绍了如何:
|
|
使用加速组织病理学技术节省时间对于快速识别和治疗疾病至关重要。
WSI I/O 和图像处理方面的挑战
将深度学习整合到整个幻灯片图像的处理中需要的不仅仅是训练和测试模型。使用深度学习的图像分析需要大量的预处理和后处理来改进解释和预测。必须为建模准备整个幻灯片图像,并且生成的预测需要额外的处理才能解释。示例包括伪影检测、颜色归一化、图像子采样和删除错误的预测。
此外,整个幻灯片图像的尺寸通常非常大,分辨率高于 100,000 x 100,000 像素。这会强制平铺图像,这意味着在建模中使用整个幻灯片图像中的一系列子样本。将这些平铺图像从磁盘加载到内存中,然后处理平铺图像可能非常耗时。
加速 WSI I/O 和图像处理的工具
本文中介绍的用例使用下面详述的 GPU 加速工具进行基准测试。
cuCIM
cuCIM(Compute Unized Device Architecture Clara IMage)是一个用于多维图像的开源加速计算机视觉和图像处理软件库。用例包括生物医学、地理空间、材料和生命科学以及遥感。cuCIM 库在宽松许可证 (Apache 2.0) 下公开提供,并欢迎社区贡献。
Magnum IO GPU 直连存储
Magnum IO GPU Direct Storage (GDS) 提供存储 I/O 加速,这是用于并行、异步和智能数据中心 I/O 的 Magnum IO 库的一部分。 GDS 为 GPU 内存和存储之间的直接内存访问 (DMA) 传输提供直接数据路径,从而避免通过 CPU 的反弹缓冲区。此直接路径可增加系统带宽、减少延迟并减少 CPU 上的使用负载。
总体而言,GDS 具有以下优势:
|
GDS 可以通过 RAPIDS kvikIO 包从 Python 访问。KvikIO 提供 Python和C++ API,能够在 GDS 支持下执行读取或写入操作。(当 GDS 不可用时,回退到基本 POSIX 和操作)。(cudaMemcpy)
整张玻片图像数据 I/O
当在组织病理学中通过数字全玻片图像扫描仪对载玻片进行数字化时,以多种放大倍率拍摄高分辨率图像。WSI 具有金字塔形数据结构,每个放大倍率拍摄的图像都形成 WSI 的“层”。这些图像的最大放大倍率通常是 200 倍(使用 10 倍放大倍率的 20 倍物镜)或 400 倍(在 10 倍放大率下使用 40 倍物镜)。
图 1 和图 2 显示了乳腺癌研究的基准用例比较。图1显示了H&E染色数字病理学载玻片的缩小视图,其中突出显示了感兴趣区域(ROI)。图 2 显示了与图 1 中突出显示的 ROI 相对应的高分辨率视图。
为此用例选择了具有以下特征的图像数据集:
|

图1.来自乳腺癌研究的典型全玻片图像,感兴趣区域以黑色突出显示。对应于各个 TIFF 图块边界的网格线以灰色叠加。
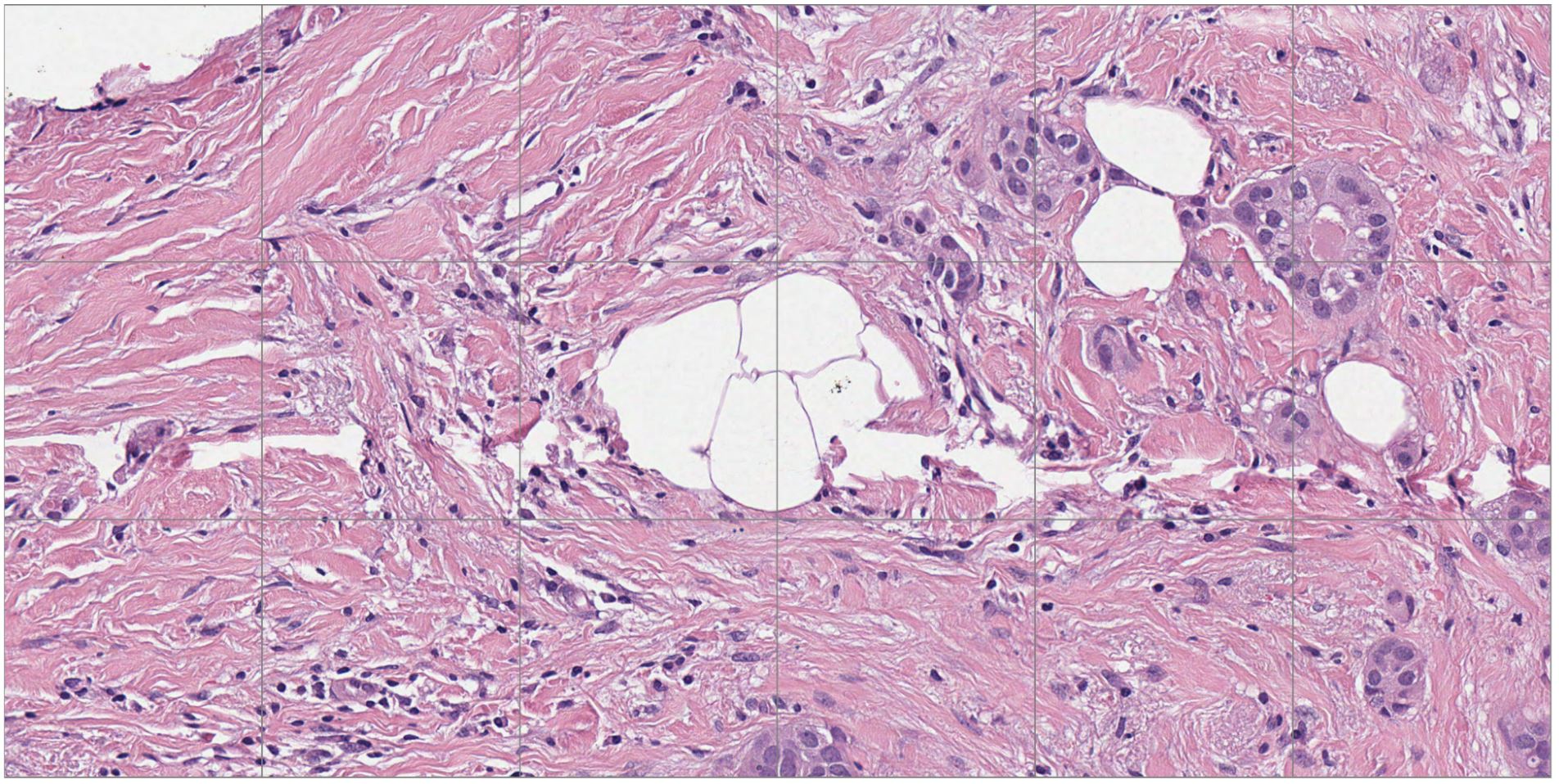
图2.图 1 中黑色矩形突出显示的区域的高分辨率视图
数字病理学用例
本节介绍三种不同的数字病理学用例。
用例 1:将 WSI 图像加载到 GPU 阵列中
在此图像分析用例中,磁盘中 WSI 图像中的每个单独切片都加载到具有和不具有 GDS 的 1D GPU 缓冲区中。目标 GPU 阵列已预先分配。在每次使用中,读取图像都会被重塑并放置在 GPU 输出数组中的适当位置,如图 3 所示。
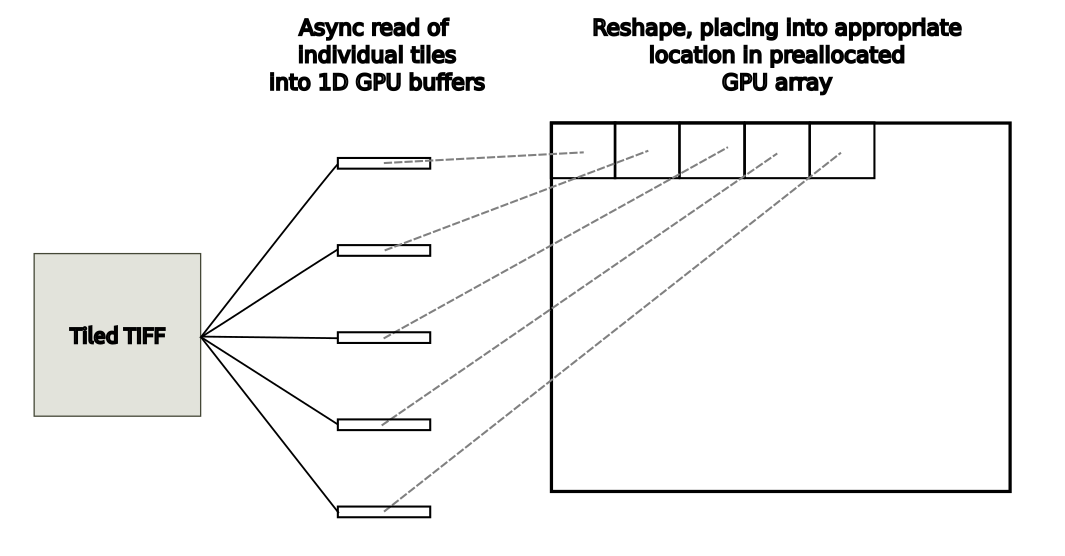
图3.从主机上的平铺 TIFF 图像读取到 GPU 阵列
图 4 中所示的基准测试结果绘制了相对于使用加速,然后调用 to 将阵列从主机传输到 GPU。在没有启用 GDS 的情况下,kivikIO 为单线程和并行读取场景提供了 2.0 倍和 4.2 倍的加速。启用 GDS 后,单线程机箱的加速度从 2.0 倍提高到 2.7 倍,而并行读取从 4.2 倍提高到 11.8 倍。(tifffile.imreadcupy.asarray)
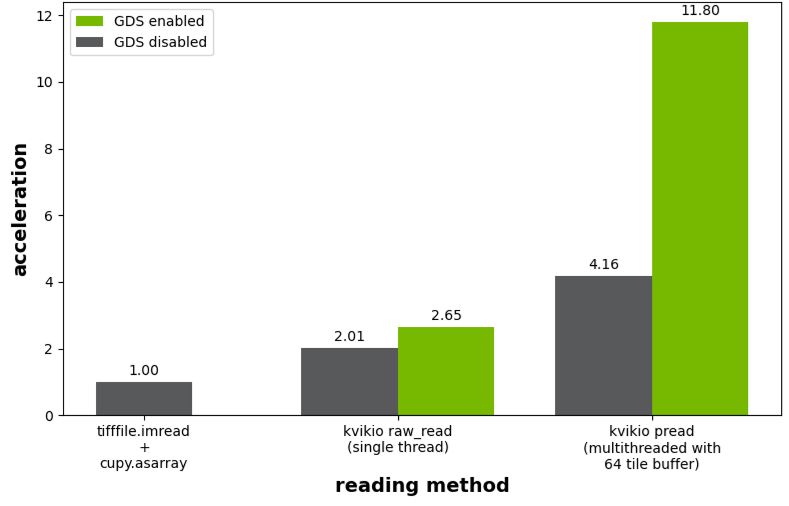
图4.平铺 TIFF 图像读取相对于使用 tifffile.imread 后跟 cupy.asarray 的性能
用例 2:写入未压缩的 Zarr 文件
在此图像分析用例中,从 GPU 内存中的 CuPy 数组开始。例如,您可以从用例 1 的输出开始。然后将此 GPU 阵列平铺写入具有和不使用 GDS 的单独 Zarr 文件中。这涉及将 6,084 MB 的数据写入磁盘,其中每个“块”都写入一个独立的文件。各种区块形状的文件大小为:
|
图 5 显示了块大小(2048、2048、3)的结果。对于除最小块大小(256、256、3)之外的所有区块大小,启用 GDS(未显示)都有实质性的好处。
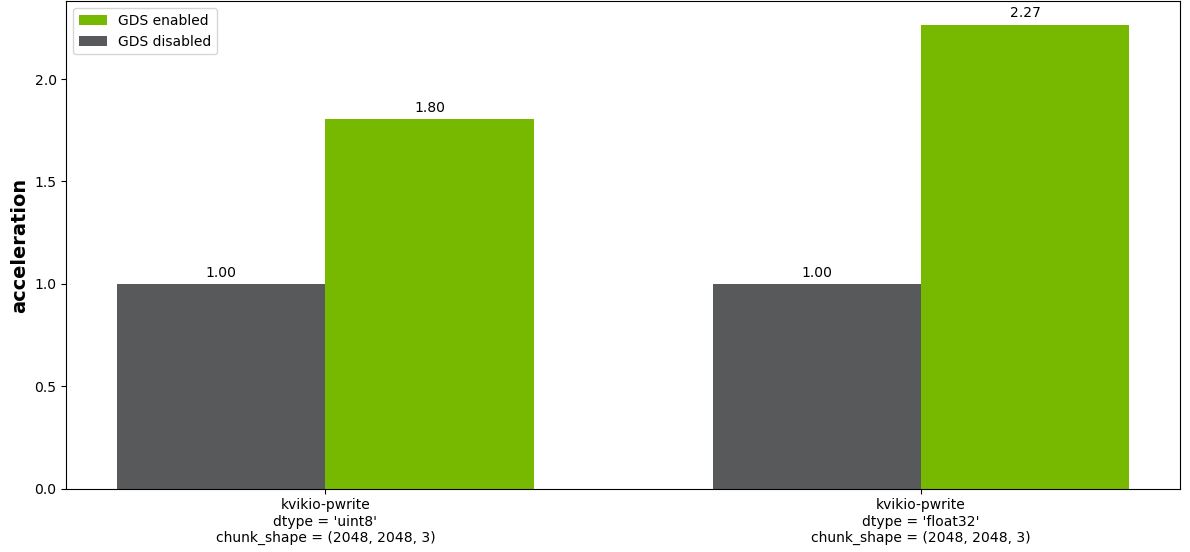
图5. GDS 写入 Zarr 文件(分块数组)的相对性能。将显示 8 位整数和 32 位浮点图像的结果。
用例 3:平铺图像处理工作流
此图像分析用例将加载、处理和保存整个幻灯片图像组合到一个应用程序中。此用例如图 6 所示。步骤如下:
|
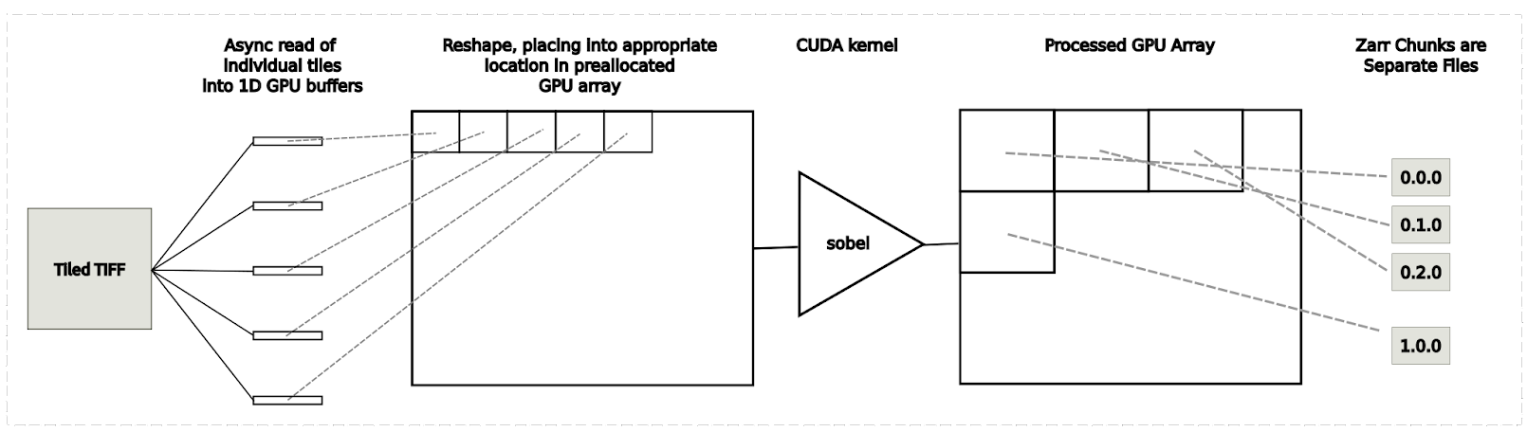
图6.用于平铺处理的内存密集型“全局”方法的工作流
一种方法是从用例 1 执行平铺读取,然后将 CUDA 内核应用于整个阵列,并从用例 2 执行平铺写入。此方法的 GPU 内存开销相对较高,因为完整映像的两个副本必须存储在 GPU 内存中。在图 8 所示的基准测试结果中,此方法标记为“多线程全局”。
一种更节省内存的方法是并行异步读取、处理和写入各个磁贴。在这种情况下,GPU 内存要求大大降低,因为任何时候只有一小部分图像在 GPU 内存中。此方法如图 7 所示。在图 8 所示的基准测试结果中,此方法标记为“多线程平铺”。
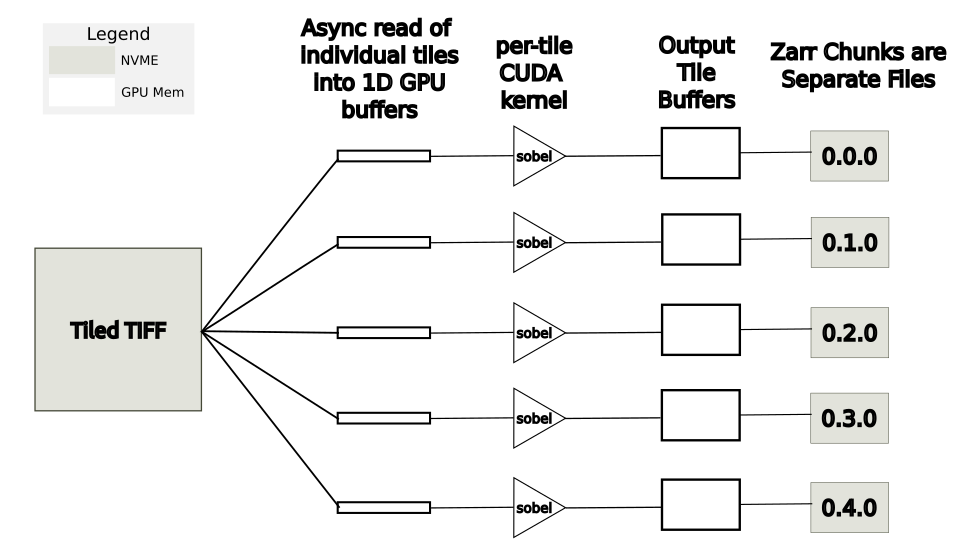
图7.切片处理的低内存方法
与禁用 GDS 的多线程全局方法相比,多线程平铺方法的结果在性能方面进行了规范化。如果没有 GDS,执行异步平铺处理会损失 15% 的性能,但随着 GDS 运行时提高 ~2 倍,比使用 GDS 的全局方法略快。
按平铺 CUDA 处理方法的主要缺点是,由于平铺处理,简单实现当前无法处理潜在的边框伪影。对于像素计算(如色彩空间转换或数字病理学中从RGB到吸光度单位的转换)而言,这与无关紧要。
但是,对于涉及卷积的操作,使用单个切片的边缘扩展而不是来自相邻切片的实际数据可能会产生细微的伪影。为了处理类似的图像分析用例,我们建议首先保存到 Dask 数组,然后使用地图块执行平铺处理,这可以考虑此类边界因素。
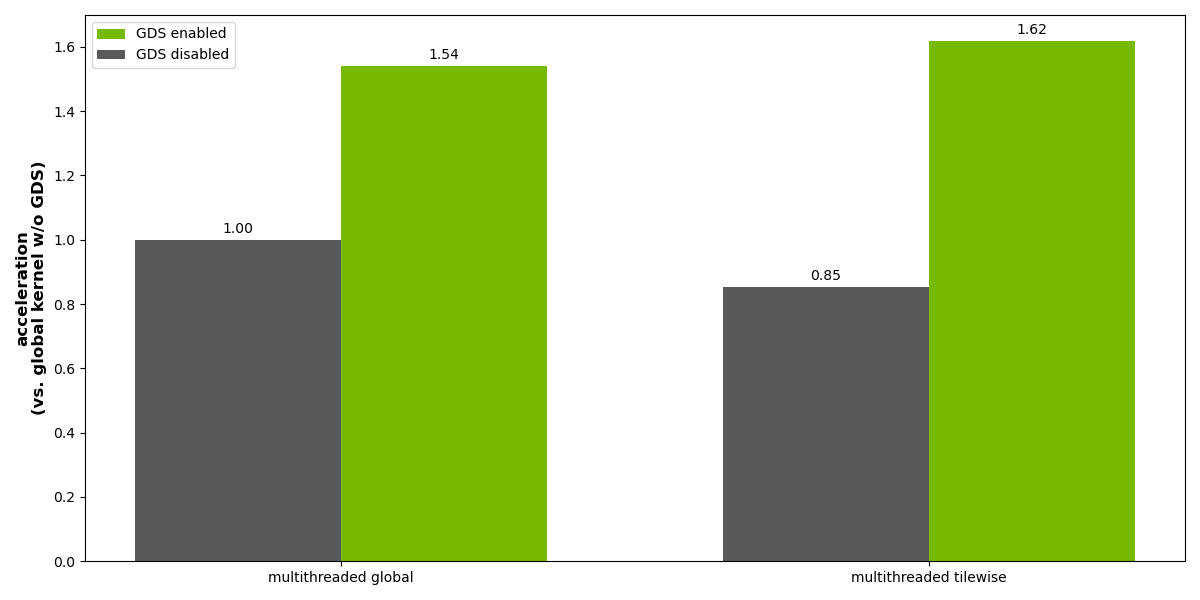
图8. 使用多线程全局方法(左)和多线程平铺方法(右)进行处理的基准测试结果。加速度相对于没有 GDS 的全局处理的情况显示。
用于加速 WSI I/O 和图像处理的示例脚本
用于基准测试的脚本可以从 cuCIM 存储库的 examples/python/gds_whole_slide 文件夹中获取。目前,这些图像分析用例正在使用 RAPIDS kvikIO 进行 GDS 加速读/写操作。这些组织病理学演示仅用于说明目的,尚未针对生产用途进行测试。将来,我们希望扩展 cuCIM API,以支持执行这些类型的平铺读写操作的方法。
摘要:为什么加速 WSI I/O 很重要
本文中介绍的图像分析用例表明,NVIDIA GPU 直连存储对于减少需要读取和写入平铺数据集的各种高分辨率图像的 I/O 时间具有实质性的好处。使用加速组织病理学技术所显示的节省时间对于快速识别和治疗疾病至关重要。我们期待在未来扩展未压缩的TIFF和Zerr数据的这些初步结果,以包括压缩数据。在此期间,我们邀请您使用Conda或PyPI下载cuCIM。GDS 是 CUDA 11.8 的一部分。有关安装要求,请参阅 GDS 文档。
作者简介
 关于 Greg Lee Greg Lee 是 NVIDIA Clara 團隊的軟體工程師。他在密歇根大学获得生物医学工程博士学位,并在磁共振成像(MRI)研究领域工作多年。由于对GPU加速计算感兴趣,他是 RAPIDS cuCIM 的共同创建者,也是 CuPy 的频繁贡献者。他还参与了几个开源科学 Python 库(PyWavelets,SciPy,scikit-image和Zarr)的开发和维护。
关于 Greg Lee Greg Lee 是 NVIDIA Clara 團隊的軟體工程師。他在密歇根大学获得生物医学工程博士学位,并在磁共振成像(MRI)研究领域工作多年。由于对GPU加速计算感兴趣,他是 RAPIDS cuCIM 的共同创建者,也是 CuPy 的频繁贡献者。他还参与了几个开源科学 Python 库(PyWavelets,SciPy,scikit-image和Zarr)的开发和维护。
 关于 Rahul Choudhury Rahul 是 NVIDIA 医疗保健部门的高级软件经理。他领导 Clara 应用团队,专注于从医学图像中提取意义。Rahul 热衷于医学图像可视化、人工智能和设计思维交叉领域的创新。他在医学成像领域的经验涵盖了实时成像和 3D 模式后处理的细分市场。Rahul 拥有加利福尼亚州斯坦福大学的生物医学信息学硕士学位。
关于 Rahul Choudhury Rahul 是 NVIDIA 医疗保健部门的高级软件经理。他领导 Clara 应用团队,专注于从医学图像中提取意义。Rahul 热衷于医学图像可视化、人工智能和设计思维交叉领域的创新。他在医学成像领域的经验涵盖了实时成像和 3D 模式后处理的细分市场。Rahul 拥有加利福尼亚州斯坦福大学的生物医学信息学硕士学位。
 关于 Gigon Bae Gigon Bae 是 NVIDIA 的软件工程师,致力于 NVIDIA Clara,这是一个用于人工智能成像、基因组学以及智能传感器开发和部署的医疗保健应用程序框架。他之前曾在相机团队担任 NVIDIA Jetson/Shield 平台上 Android/Linux 相机模块的相机软件工程师。 在 2015 年加入 NVIDIA 之前,他获得了韩国科学技术院 (KAIST) 的博士学位,专攻软件测试和程序分析,重点是将自动测试用例生成技术应用于 GUI 软件和单元级代码,以及实证研究。
关于 Gigon Bae Gigon Bae 是 NVIDIA 的软件工程师,致力于 NVIDIA Clara,这是一个用于人工智能成像、基因组学以及智能传感器开发和部署的医疗保健应用程序框架。他之前曾在相机团队担任 NVIDIA Jetson/Shield 平台上 Android/Linux 相机模块的相机软件工程师。 在 2015 年加入 NVIDIA 之前,他获得了韩国科学技术院 (KAIST) 的博士学位,专攻软件测试和程序分析,重点是将自动测试用例生成技术应用于 GUI 软件和单元级代码,以及实证研究。
相关贴子
-
 技术分享
技术分享什么是 OCP 3.0 NIC?
2025.07.25 30分钟阅读 -
 技术分享
技术分享选 CPU 看核心数量还是时钟速度?
2024.10.11 27分钟阅读 -
 技术分享
技术分享多站点分布式 GPU 架构:探索本地分布式 GPU 资源
2025.12.12 49分钟阅读






